申请日2014.09.05
公开(公告)日2014.12.24
IPC分类号C12Q1/68; C12Q1/04; C12Q1/06
摘要
本发明公开一种检测废水系统中氨氧化菌群落结构和丰度的方法,具体步骤包括:(A)提取废水系统活性污泥中微生物的全部基因组DNA;(B)以全部基因组DNA为模板,以序列号为SEQIDNo.1的amoA1F和序列号为SEQIDNo.2的amoA2R为引物进行PCR扩增;(C)以罗氏454焦磷酸测序法对扩增产物进行测序,对测序结果运用Mothur程序划分操作分类单元,选择操作分类单元中具有99%相似度的代表性序列与数据库GenBank中公开序列进行在线BLASTn比对,根据比对结果划分系统进化树,得到氨氧化菌的群落结构分布和丰度;本发明操作简单、成本低廉、检测迅速、序列分析准确度高,能够定性分析氨氧化菌的不同群落结构组成,定量分析氨氧化菌不同种属的丰度,并且可同时对多个样品分析,具有较强的实用性,易于推广应用。
权利要求书
1.一种检测废水系统中氨氧化菌群落结构和丰度的方法,其特征在于,具体步骤如下:
A)提取废水系统活性污泥中微生物的全部基因组DNA;
B)PCR扩增,以全部基因组DNA为模板,以序列号为SEQ ID No.1的amoA1F和序列号为SEQ ID No.2的amoA2R为引物进行PCR扩增;
C)以罗氏454焦磷酸测序法对扩增产物进行测序,对测序结果运用Mothur程序划分操作分类单元,选择操作分类单元中具有99%相似度的代表性序列进行BLASTn比对,根据比对结果划分系统进化树,即获得废水系统中氨氧化菌群落结构和丰度检测结果。
2.根据权利要求1所述检测废水系统中氨氧化菌群落结构和丰度的方法,其特征在于,步骤B中,PCR反应体系:10×buffer 3μL,MgCl2 1.8μL,浓度为5U/μL的Taq酶 0.2 μL,dTNP1.4μL,引物SEQ ID No.1和SEQ ID No.2各0.3μL;DNA模板2μL;无菌双蒸水21μL;
PCR反应条件:94℃预变性10min;94℃变性45s,55℃退火45s,72℃延伸1min,共35个循环;最后72℃下延伸4min。
说明书
一种检测废水系统中氨氧化菌群落结构和丰度的方法
技术领域
本发明涉及生物技术领域,特别是一种检测分析废水系统中氨氧化菌群落结构和丰度的方法。
背景技术
随着生活水平的提高和工业的迅速发展,废水排放量逐渐升高,废水中氮含量逐年上升,如何能有效去除废水中的氮,是降低废水氮元素污染,减轻水体富营养化的关键措施,现阶段,针对废水脱氮过程中脱氮微生物的研究逐渐成为热点,废水生物脱氮系统主要包括硝化和反硝化过程,其中硝化过程主要微生物为硝化菌,包括氧化氨盐为亚硝酸盐的氨氧化菌和氧化亚硝酸盐为硝酸盐的亚硝酸盐氧化菌,氨氧化菌是废水生物脱氮过程中的关键微生物,决定着废水中亚硝酸盐的含量和浓度,直接影响后续硝化反硝化、短程硝化反硝化等过程的效率,是生物脱氮过程的第一环节,也是关键步骤,氨氧化菌在废水生物处理系统中群落结构和种属丰度是保证废水生物脱氮能力长期稳定的首要条件,是影响废水处理系统出水氨氮浓度的关键因素。
在废水生物系统中,微生物菌群是由多种微生物构成、具有一定空间分布的聚集体,这些微生物相互依存、相互竞争,具有复杂的种群关系,当进行菌种分离培养后,将无法获取自然条件下微生物菌群结构与空间分布的信息,其次,由于微生物的基因表达和代谢活性受外界环境因素的影响较大,菌种的分离培养将会改变微生物的生理生化特性,从而降低对实际工程实践的指导意义。此外,纯培养需要较长的时间,而且有些菌种( 如硝化菌中的Nitrospira) 目前还无法通过纯培养技术实现纯种分离,即无法进行分离培养,因此分子生物学的分析方法成为大部分功能菌群的研究手段,如变性梯度凝胶电泳(DGGE)、荧光原位杂交(FISH)、实时荧光定量PCR(real time Q-PCR)等均为研究分析废水生物系统活性污泥群落结构的常用手段。
目前,DGGE分析方法是对氨氧化菌的定性分析主要手段,如《污水人工快速渗滤系统中氨氧化菌16S rDNA的DGGE分析》(马鸣超等,高校地质学报,2007年第13卷第4期)中章根据DGGE图谱中条带数量和变化,定性分析了污水处理系统中氨氧化菌的群落结构组成,但其在氨氧化菌的定性分析中,采用的DGGE技术无法确定氨氧化菌群落中具体种属,分析过程存在很大偏差,难以准确分析氨氧化菌的不同群落结构组成。
此外,针对氨氧化菌的定量分析主要是FISH技术和荧光实时定量PCR技术,其中,FISH技术是运用与荧光素结合的寡聚核苷酸探针和核酸进行碱基配对杂交,然后通过荧光系统检测对样品DNA定量分析,如《OLAND生物脱氮系统运行及其硝化菌群的分子生物学检测》(张丹等,应用与环境微生物学报,2003年第9卷第5期)一文运用FISH技术定量分析氨氧化菌的群落结构组成变化,但FISH技术存在对探针要求高、操作复杂,对短链cDNA探针效率明显下降,无法达到100%的杂交效率等缺点,难以对全部的氨氧化菌群落结构组成做定量分析。
荧光实时定量PCR技术是在PCR反应体系中加入荧光基团,在经热循环后检测荧光累积量,再与标准曲线比对来定量分析样品DNA,专利号为CN 201110305190.4,发明名称“活性污泥中氨氧化菌实时荧光定量PCR检测方法”一文中,以实时荧光定量PCR技术定量检测氨氧化菌的群落结构;荧光实时定量PCR技术虽然应用于氨氧化菌的分析,但是该技术操作难度大,需要制备标准品DNA,在DNA定量分析中标准曲线不统一,结果可比性差,且实验成本较高,难以对氨氧化菌的群落结构做可靠、快速的定量检测分析。
由于以上不同分析技术存在各自的缺点,提供一种简单、快速、有效检测废水处理系统中氨氧化菌的群落结构检测丰度变化的方法,一直是本领域亟待解决的问题。
发明内容
针对上述问题,提供检测废水系统中氨氧化菌群落结构分布和丰度的方法,可以实现简单、快速检测废水生物处理系统中氨氧化菌群落结构和种属丰度检测分析,本发明是这样实现的:
一种检测分析废水系统中氨氧化菌群落结构和丰度的方法,具体步骤如下:
A)提取废水生物处理系统活性污泥中微生物的全部基因组DNA;
B)PCR扩增,以全部基因组DNA为模板,以序列号为SEQ ID No.1的amoA1F和序列号为SEQ ID No.2的amoA2R为引物进行PCR扩增。
C)以罗氏454焦磷酸测序技术对扩增产物进行测序,测序数据结果运用Mothur程序拆分、降噪和去嵌合体,并划分操作分类单元(OTUs)。选择操作分类单元(OTUs)中具有99%相似度的代表性序列与数据库GenBank中公开序列进行在线BLASTn比对,根据比对结果划分系统进化树,得到氨氧化菌的群落结构分布和丰度检测分析结果。
优选的,本发明所述检测分析废水系统中氨氧化菌群落结构和丰度的方法,步骤B中PCR反应体系:10×buffer 3μL,MgCl21.8μL,浓度为5U/μL的Taq酶 0.2 μL,dTNP1.4μL,引物SEQ ID No.1和SEQ ID No.2各0.3μL;DNA模板2μL;无菌双蒸水21μL;
PCR反应条件:94℃预变性10min;94℃变性45s,55℃退火45s,72℃延伸1min,共35个循环;最后72℃下延伸4min。
本发明提供通过特异性引物amoA1F和amoA2R,实现对氨氧化菌的功能基因氨单加氧酶基因(amoA)特异性扩增,然后将扩增产物进行罗氏454 FLX焦磷酸测序,测序结果通过生物多样性分析和BLASTn比对,即可获得氨氧化菌的群落结构组成和种属丰度;本发明操作简单、成本低廉、检测迅速、序列分析准确度高,能够定性分析氨氧化菌的不同群落结构组成,定量分析氨氧化菌不同种属的丰度,并且可同时对多个样品分析,能够应用于废水生物处理系统中氨氧化菌的检测分析和实时监控,保证废水脱氮效果;也可应用于天然水体环境中氨氧化菌的检测和定量分析,具有较强的实用性,易于推广应用。
具体实施方式
以下通过具体实施例对本发明作进一步说明:
实施例1 设计氨单加氧酶基因(amoA)特异性引物
为保证扩增过程准确,扩增产物单一,引物设计方法选用在线Primer-BLAST引物设计,其中登录网址为http://www.ncbi.nlm.nih.gov/tools/primer-BLAST。
选取amoA基因模板序列FASTA文件,设置最佳的引物设计参数,并同时对每个引物BLAST分析,使引物具有特异性。设计获得的引物运用软件Oligo7.0进行评估筛选,最后得到的特异性引物,即序列号为SEQ ID No.1的amoA1F和序列号为SEQ ID No.2的amoA2R,如下所示:
引物 序列 amoA1F 5'-TCATGGACACCGTCTTGCTG-3' amoA2R 5'-CCAAAGGTTCGCAATGAGCC-3'
实施例2 某城市污水处理厂活性污泥中氨氧化菌的群落结构和种属丰度分析
1、总基因组DNA的提取
样品取某城市污水处理厂活性污泥,采用美国MP Biomedicals公司生产的FastDNA 土壤自旋提取试剂盒对活性污泥样品提取总基因组DNA,操作严格按照说明书进行,利用ND-1000紫外分光光度计检测总基因组DNA的浓度和纯度。
2、 amoA基因PCR扩增
配置PCR反应体系为30μL。首先根据样品计算好各组成成分含量,然后把Taq酶、MgCl2、正反向引物、脱氧核糖核苷酸dTNP和无菌双蒸水依次加入1.5mL离心管中,低速离心使其混合均匀。把上述反应体系分装到各个PCR反应管中,然后加入样品DNA模板,并低速离心混合均匀。
amoA基因特异性扩增体系如下:
组分 体积μL 无菌双蒸水/μL 21 脱氧核糖核苷酸dTNP/μL 1.4 正向引物(10mM)/μL 0.3 反向引物(10mM)/μL 0.3 10×buffer 3 MgCl2 1.8 Taq酶(5U/μL)/μL 0.2 DNA模板/μL 2 总体积/μL 30
PCR扩增条件为:变性94℃下10min;35个循环反应,每个循环包括94℃45s,55℃45s,72℃1min;最后72℃下延伸4min。
3. 罗氏454 FLX焦磷酸测序和数据分析
将步骤2获得的PCR扩增产物用Takara切胶纯化试剂盒进行切胶纯化,严格按照试剂盒说明书进行,纯化后的样品溶解在适量的TE缓冲液中,将纯化后的DNA样品进行罗氏454 FLX焦磷酸测序。
罗氏454FLX焦磷酸测序数据结果首先采用Mothur程序对序列进行拆分、降噪和去嵌合体,并以97%的相似度划分操作分类单元(OTUs)。从每个OUT中选取具有99%相似度的代表性序列与数据库GenBank中公开序列进行在线BLASTn比对。
选择某污水处理厂活性污泥样品中每个OTU的代表性序列进行BLASTn比对,部分比对结果如图1所示。
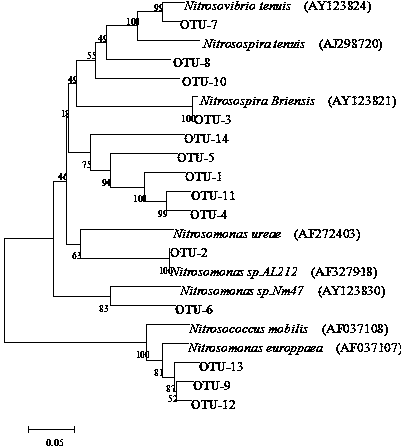
根据比对结果,选择相似度95%以上,且有文献报道的已经分离鉴定出的序列,利用MEGA6.0软件构建系统进化树。系统进化树采用邻位相连法,进化树节点处Bootstrap值设定为1000,进化距离为0.05,其结果如图2所示,根据图2可以确定氨氧化菌群主要属于Nitrosospira属和Nitrosomonas属,在Nitrosospira属中主要是Nitrosospira Briensis、Nitrosospira tenuis,在Nitrosomonas属中主要是Nitrosomonas ureae、Nitrosomonas europpaea、Nitrosomonas sp.Nm47。
根据活性污泥样品中amoA基因的不同OTUs,做amoA基因OTUs的丰度和分布图,定量分析不同的氨氧化菌群,其结果如图3所示,amoA基因中主要是OUT-6、OUT-7和OUT-9,而它们分别与Nitrosomonas sp.Nm47、Nitrosovibrio tenuis和Nitrosomonas europpaea有较大的相似度,由此可以检测判断出该污水处理厂活性污泥中的氨氧化菌的群落结构分布和种属丰度。
以上实施例仅用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改,其均应涵盖在本发明的权利要求范围当中。